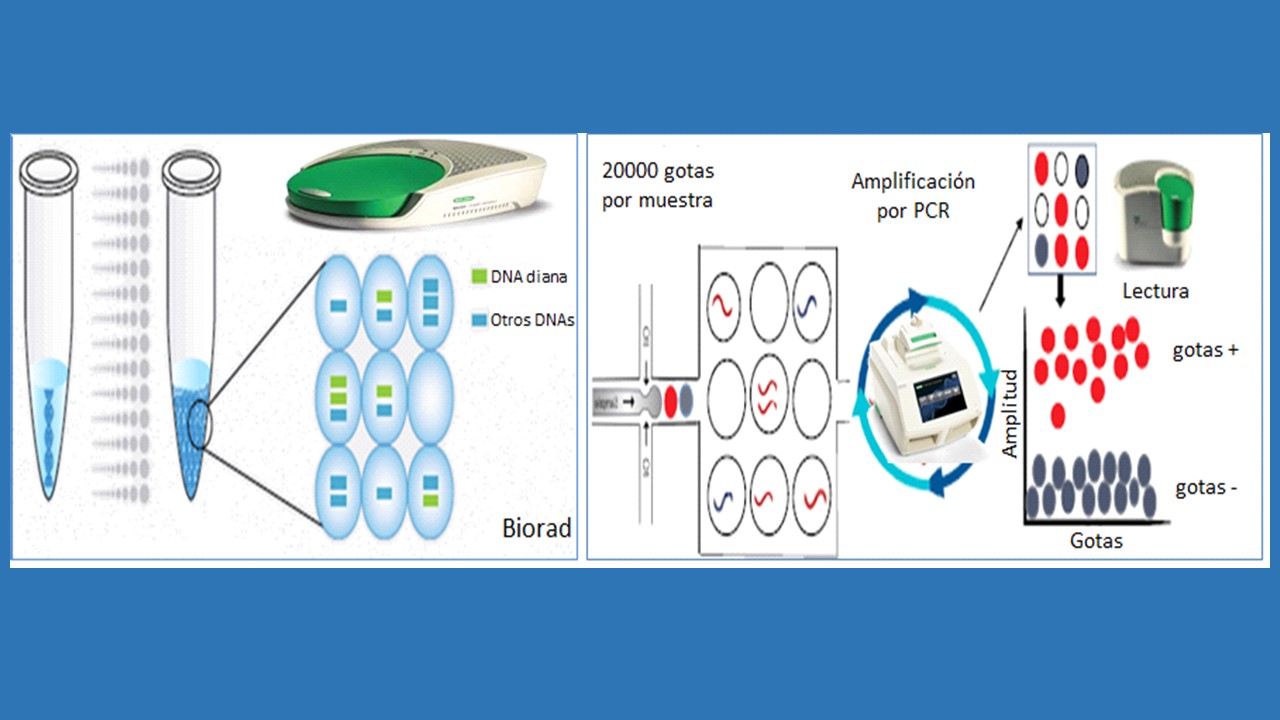
La paratuberculosis bovina (PTB) es una enfermedad infecciosa causada por Mycobacterium avium subsp. paratuberculosis (MAP) que afecta a rumiantes silvestres y domésticos y que representa un grave problema económico para las granjas de ganado bovino. Aunque la vacunación se ha visto que es la mejor herramienta para el control de esta enfermedad su utilización no está permitida en la mayoría de los países, por lo que en la actualidad el control de esta enfermedad se basa en la eliminación de los animales infectados y en la mejora de las condiciones higiénico-sanitarias y de bioseguridad de las granjas. Las técnicas de diagnóstico más utilizadas son el ELISA para la detección de anticuerpos frente a MAP y la PCR a tiempo real (qPCR) para la detección de DNA de MAP en muestras de heces. Sin embargo, estos métodos presentan muy baja sensibilidad para la detección de animales en las fases iniciales de la infección, debido a que la carga bacteriana no es tan abundante como en las muestras de animales con signos clínicos y a que la eliminación de MAP a través de las heces es intermitente. Por ello, es necesario desarrollar nuevas tecnologías capaces de detectar y cuantificar MAP con mayor sensibilidad. La ddPCR (por sus siglas en inglés, droplet digital polymerase chain reaction) es un tipo de PCR de nueva generación que permite la detección y cuantificación de un número bajo de copias de DNA con una alta reproducibilidad. Esta tecnología primero divide la muestra original en miles de microgotas, de manera que cada microgota puede o no contener el DNA diana. El siguiente paso es llevar a cabo una qPCR convencional, y finalmente leer las microgotas una a una mediante un lector de ddPCR capaz de clasificar cada microgota como positiva o negativa en función de si tiene el DNA diana amplificado o no. Además, la tecnología de ddPCR permite la cuantificación del DNA diana haciendo uso de la fracción de microgotas positivas frente al número de microgotas totales. La ddPCR se ha usado anteriormente para el diagnóstico de otras enfermedades causadas por micobacterias, como la tuberculosis bovina y humana, y también ha sido utilizada para la cuantificación de DNA genómico extraído de cultivos bacteriológicos de MAP con fines analíticos, pero no se había usado anteriormente para la detección de la infección por MAP en muestras de sangre y heces de animales infectados en condiciones naturales. El Departamento de Sanidad Animal de NEIKER en colaboración con el SERIDA y con el departamento de Conservación de Recursos Naturales de NEIKER ha diseñado y evaluado un protocolo de ddPCR para la detección y cuantificación de DNA de MAP en muestras de heces y sangre. Para ello, se han utilizado como controles DNAs extraídos de muestras de animales sin lesiones histológicas compatibles con PTB o provenientes de granjas negativas a ELISA y a qPCR fecal en al menos tres años consecutivos; y como casos, muestras de animales con lesiones asociadas a la PTB. Los resultados obtenidos con la ddPCR se compararon con los resultados obtenidos con otras técnicas de diagnósticos como el ELISA, la qPCR y el cultivo bacteriológico de muestras de heces y tejidos intestinales. Los resultados obtenidos con la ddPCR de muestras fecales fueron similares a los obtenidos con los tests diagnósticos convencionales, detectándose mayoritariamente animales con lesiones difusas, el tipo de lesión más grave. Por otro lado, la ddPCR de muestras de sangre detectó en su mayoría animales con lesiones focales (sensibilidad 70 %, especificidad 100%), el tipo de lesión menos grave y generalmente asociado a infecciones subclínicas. Además, los resultados obtenidos con la tecnología de ddPCR en muestras de sangre se correlacionaron con los obtenidos con un ELISA específico para la detección en suero del biomarcador ABCA13, un marcador previamente validado para la detección de animales en fase subclínica y con lesiones focales (http://www.blogsanidadanimal.com/nuevos-elisas-para-el-diagnostico-de-la-paratuberculosis-basados-en-la-deteccion-de-biomarcadores-bovinos/). Este estudio ha sido publicado en Frontiers in Veterinary Science y se puede consultar en este enlace. (Link: https://www.frontiersin.org/articles/10.3389/fvets.2022.944189/full).
Este estudio ha sido financiado por el Ministerio de Ciencia, Innovación y Universidades (Fondos Europeos para el Desarrollo Regional (FEDER, Una manera de hacer Europa) y fondos regionales (PCTI 2021-2023 (GRUPIN: IDI2021-000102). María Canive, Gerard Badía Bringué y Cristina Blanco-Vázquez son beneficiarios de becas predoctorales del MCIN (MCIN/AEI/10.13039/501100011033 y FSE Invierte en tu futuro); FPI2016-00041, PRE2019-090562, y CPD2016-0142, respectivamente.
Imagen: Generación de microgotas, amplificación por PCR y lectura clasificación de gotas en positivas y negativas. Imagen de la derecha (Biorad), izquierda (Nyaruaba et al., 2019).